敲除sgRNA“保姆级”设计教程
前面小编介绍了利用CRISPR/Cas9技术获取基因敲除细胞株的实验流程,本期内容小编将为小伙伴们带来敲除sgRNA设计的“保姆级”教程。每一种Cas蛋白均可在基因组上识别其特有的原间隔序列临近基序(PAM),并在sgRNA的引导下在基因组PAM基序上游对基因组dsDNA进行切割。每种Cas蛋白识别的PAM基序不同,例如运用最广泛的spCas9的PAM序列为NGG。部分不同物种来源的Cas9酶d对应的PAM基序如表1。
表1.不同Cas9酶PAM序列列表

注:N为任意核苷酸;W为A/T;R为A/G;H为A/T/C;Y为T/C;V为A/C/G;
高编辑效率的gRNA通常需要满足以下设计原则:
1. sgRNA的长度:SpCas9的sgRNA一般为20nt,SaCas9的sgRNA长度为21nt;
2. sgRNA序列应尽可能覆盖最多的靶标基因转录本:针对多转录本的共有序列进行设计;
3. sgRNA序列的碱基组成:基因特异的sgRNA 模板序列位于PAM基序之前,sgRNA的序列应避免以4个以上的T结尾,GC含量最佳40%-60%;
4. 如果构建U6启动子或T7启动子驱动sgRNA的表达载体,需要考虑sgRNA的5'碱基为G或GG,来提高其转录效率;
5. 设立在独立外显子上,尽量靠近蛋白N端位置(编码区的前1/3)。若想要造成基因移码突变,需要尽量靠近基因编码区的ATG下游,最好位于第一或第二外显子上,可增加基因敲除效率。
表2. sgRNA常用设计网站推荐

注:这些网站都是目前sgRNA设计的常用网站,采用的算法不同,各有优缺点。针对同一个基因的敲除,小伙伴们可以多使用几个网站进行sgRNA设计,对结果进行比对,根据设计原则择优选取。
下面小编以敲除人源 NLE1 基因为例,利用CRISPOR网站进行特异性sgRNA设计:
1、打开NCBI,选择Gene,搜索基因名称及物种名称:

2、下拉至“Gennomic regions,transcripts,and products”模块,查看转录本情况。如图,该基因共有两个转录本,深绿色方块代表外显子,两个转录本的共有CDS区为右边红方框圈起来的区域,为第二个转录本的完整编码区。
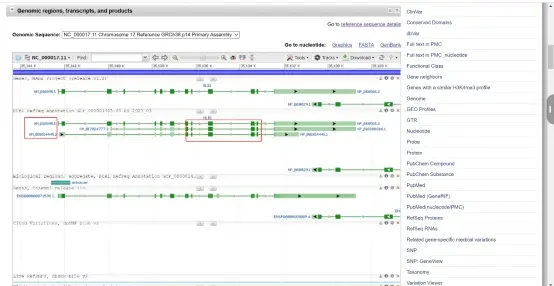
3、复制第二个转录本的NM号,用SnapGene打开,可显示编码区所在外显子的情况。中间有间隔的为不同的外显子。

4、点击共有CDS所在的第二个完整外显子,复制序列。打开CRISPOR网站→将序列复制至序列粘贴区→选择物种/基因组→选择适配Cas酶,点击“submit”,即可生成结果网页。

5、此处可显示特异性,绿色、黄色及红色分别表示在基因组上高、中、低的特异性。

此处显示sgRNA具体的序列及相关参数,主要参考MIT/CFD特异性参数:越大脱靶概率越低,建议选择这两项均在70以上的sgRNA。另外预测有效性值越大越好。最后结合sgRNA设计原则进行综合选择。

6、选定sgRNA后,复制序列→打开NCBI的Blast模块→选择基因组Blast→选择物种→跳转至blast页面→粘贴序列→点击搜索→点击右上角切换到传统视图。


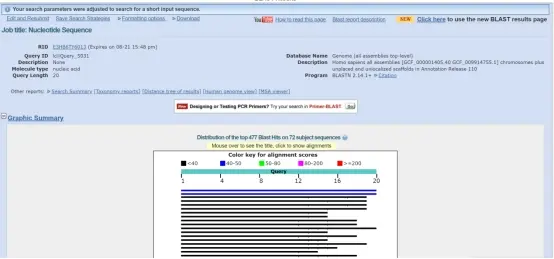
7、下拉至详细描述页,可看到完全匹配的为靶标基因所在的NC号,其余均出现不同程度的碱基错配现象,说明所选sgRNA在基因组上具备良好的特异性,可用于后续敲除实验。

以本示例中的人源 NLE1 基因为例,该基因的最长转录本在共有CDS之前仍包含7个外显子,仅在共有区设计一个sgRNA极大可能无法完全敲除该基因所有转录本所转录翻译的蛋白。针对这种情况,建议小伙伴们针对不同区域多设计几个sgRNA进行实验验证,检测实际敲除效率。
常用的基因敲除除了单sgRNA系统外,还有双sgRNA作用系统,设计原则及选择标准与上文所述一致,两种方法各具优缺点,具体表现如下:
单sgRNA敲除系统:在编码外显子上设计1个sgRNA,依赖细胞本身的非同源末端连接(NHEJ)机制引入或缺失碱基,造成移码突变,从最终得到的混合克隆中筛选符合实验要求的敲除阳性克隆。缺点:1、筛选鉴定复杂,无法从PCR结果直接判断敲除结果。PCR产物测序峰图解读困难,存在多重套峰的现象,需要做亚克隆分析才能获得准确的敲除结果。2、敲除效果存在不确定性,在外显子上设计sgRNA,尤其外显子较小时,容易产生新的mRNA剪切突变体,产生新的突变蛋白或者跟野生型蛋白类似的突变蛋白,导致后续蛋白检测分析结果不确定。
双sgRNA敲除系统:选择编码基数为非3倍数的外显子作为敲除区域,分别在外显子上游和下游的内含子上设计sgRNA,令两条sgRNA间的外显子序列和部分内含子序列同时被删除。其删除的编码碱基数确定,删除片段较大,只需通过PCR检测即可判断敲除结果,同时发生新的mRNA剪接突变体的概率大大降低,因而敲除效果更有保障,拿到细胞后鉴定分析也更简单。
总的来说,单sgRNA敲除系统效率高,设计简单,但后续蛋白检测风险较大,往往会出现WB敲除不干净的现象,导致敲除变成干扰;双sgRNA敲除需要两个sgRNA同时起作用,效率较低,但后续蛋白检测背景干净,结果有保证。
以上便是本期敲除sgRNA设计干货的全部内容,你学会了吗?若还是没学会,那么专业的事请专业的人来做:汉恒可提供sgRNA设计及相关病毒包装以及敲除细胞株构建服务,如有技术问题或产品订购需求,欢迎随时咨询!
参考文献:
[1] Smits AH, Ziebell F, Joberty G, Zinn N, Mueller WF, Clauder-Münster S, Eberhard D, Fälth Savitski M, Grandi P, Jakob P, Michon AM, Sun H, Tessmer K, Bürckstümmer T, Bantscheff M, Steinmetz LM, Drewes G, Huber W. Biological plasticity rescues target activity in CRISPR knock outs. Nat Methods. 2019 Nov;16(11):1087-1093. doi: 10.1038/s41592-019-0614-5.
[2] Hanna RE, Doench JG. Design and analysis of CRISPR-Cas experiments. Nat Biotechnol. 2020 Jul;38(7):813-823. doi: 10.1038/s41587-020-0490-7.
[3] Sharpe JJ, Cooper TA. Unexpected consequences: exon skipping caused by CRISPR-generated mutations. Genome Biol. 2017 Jun 14;18(1):109. doi: 10.1186/s13059-017-1240-0.

