Cytoscape可视化分析流程总结1230
Cytoscape可视化
1、cytoscape简介
2、cytoscape使用举例
3、图形美化
4、子网提取
5、String-蛋白质相互作用数据库
一、Cytoscape——简介
Cytoscape 是一个专注于开源网络可视化和分析的软件。它的核心是提供基础的功能布局和查询网络,并依据基本的数据的结合成可视化网络。Cytoscape 源自系统生物学,用于将生物分子交互网络与高通量基因表达数据和其他的分子状态信息整合在一起,其最强大的功能还是用于大规模蛋白质-蛋白质相互作用、蛋白质-DNA和遗传交互作用的分析。
通过Cytoscape,可以在可视化的环境下将这些生物网络跟基因表达、基因型等各种分子状态信息整合在一起,还能将这些网络跟功能注释数据库链接在一起。Cytoscape 的核心是网络,简单的网络图包括节点(node)和边(edge),每个节点可以是基因、miNRA或蛋白质等等;节点与节点之间的连接 (edge) 代表着这些节点之间的相互作用,包括蛋白与蛋白相互作用(pp),DNA与蛋白相互作用(pd)等。
注:Cytoscape安装前需安装Java
主页面注:版本为cytoscape_3.5.1

主窗口有以下几个成分组成:
菜单栏工具栏网络处理面板网络主视图窗口属性浏览板块(展示选择的点或边的属性和能够修改属性值)
1.菜单栏:File菜单:open(打开一个Cytoscape文件);New(建立一个新的网络,空的或已经存在的网络);Import(导入网络数据和属性);Export(输出数据和图)等Eidt菜单:Undo(撤销);Redo(重做);create/destory view (创建/撤销视图)等View菜单:Hide/Show Control Panel(打开或隐藏网络处理板块);Show Results Panel(网络浏览)等Select菜单:不同点和边选择选项;过滤器等Layout菜单:安排可视化网络,Plugins菜单:管理插件(install/update/delete)和添加已经安装的插件注:把需要的插件从网络上下载,并复制到系统盘的Cytoscape程序下的Plugins下就可以使用了。 2.工具栏:主要为菜单栏file的快捷键:打开、保存、导入(本地、数据库、表格)/导出(网络、表格、图片)以及网络主视图窗口中网络大小调整等
3.网络处理板块:network:包括所有创建的网络,可以选择相应的网络进行操作 style:属性(node/edge/network) Node:对点进行设置,包括:点的形状、颜色、大小;点边界线的类型、颜色、宽度;点标签的颜色、大小;点背景色的透明度等 Edge:对边进行设置,包括:边的类型、颜色、宽度;目标处箭头类型等。 Network:对网络整体属性进行设置,包括:背景标题等 Select:面板用于筛选符合特定标准的边4.属性浏览版块:查看node/edge/network属性
cytoscape-实例
本文将具体操作怎样用Cytoscape绘制网络图
Cytoscape所支持的数据格式:1.*.sif格式:nodeA<interaction>nodeB nodeC<interaction>nodeD …即文件分为三列,第一列和第三列是有相互作用关系的基因名或蛋白质名等,第二列是相互作用的名称*.sif格式简单,容易处理,但它不能规定每个节点的位置、大小、形状等。2. xgmml格式,它是一种xml格式,可以规定节点和边的许多信息,但也更复杂。3.*.txt格式:用tab分割的纯文本文件可以将文件设置成两列,每一列都是基因名(或蛋白质名),同一行的两个基因(或者蛋白质等)代表有互作关系;也可以加其他参数放在第三列,例如两基因调控的强弱系数
二、应用实例
本文以txt格式的数据进行演示绘制网络图网络文件:net.txt:共表达网络;共四列,前两列是gene id,第三列是共表达类别(正1/负-1),第四列是相关系数,以tab键分隔节点属性文件: 步骤:导入网络文件:file->import->network->file(net.txt)

其中不同标识代表着不同的含义

导入后

导入节点属性文件:file->import->table->file(node.txt)(此处为table而非network)注:node.txt:节点属性文件。四列,包含三种属性;第一列为gene id,与网络文件中一致,第二列为gene name(symbol),第三列为分子类型(蛋白编码基因/lncRNA),第四列为节点在网络中的度。

上方红色方框中“Target Table Data”的信息表示将导入的节点属性表与之前的网络图相关联,其中“Network Collection”选择的是我们之前导入的网络文件,其他参数默认如下,可不用修改,如为其他选项,则需要通络下拉列表重新选择。下方红色方框中“Preview”中:gene,name,molecular type,degree第一列gene,设置为“Key”,保证gene id不重复,第二三四列均为属性“Attribute”,如需修改,同样点击名称右侧的三角形标志。点击确定后,乍看感觉图形没有变化,但此时下方的Table Panel中已自动多出了molecular type,degree两列
可以通过style中进行简单网络图格式设置

得到网络图:

也可以自行拖拽进行微调导出文件:数据的导出可以是网络文件,表格文件或者是图片文件,图片文件包括多种图片格式以及pdf格式,在工具栏中对应选择即可点击菜单栏的图片导出*.pdf(同样可以采用export导出其它格式)

注:注意调整网络图后再保存,否则会出现网络图不完整
三、网络美化
应用上篇所得到的网络图进行美化
1.统一模板美化:
采用模板进行简单美化

得到网络图

2.根据数据含义进行美化
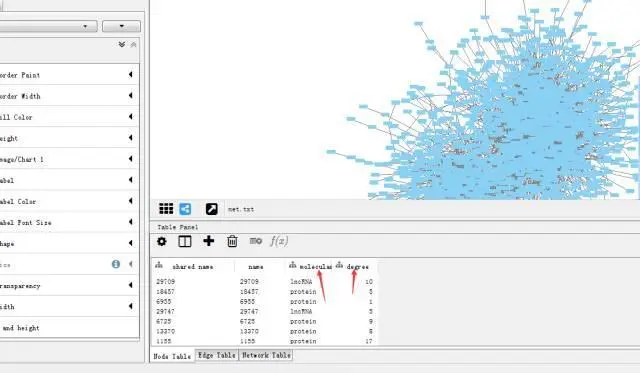
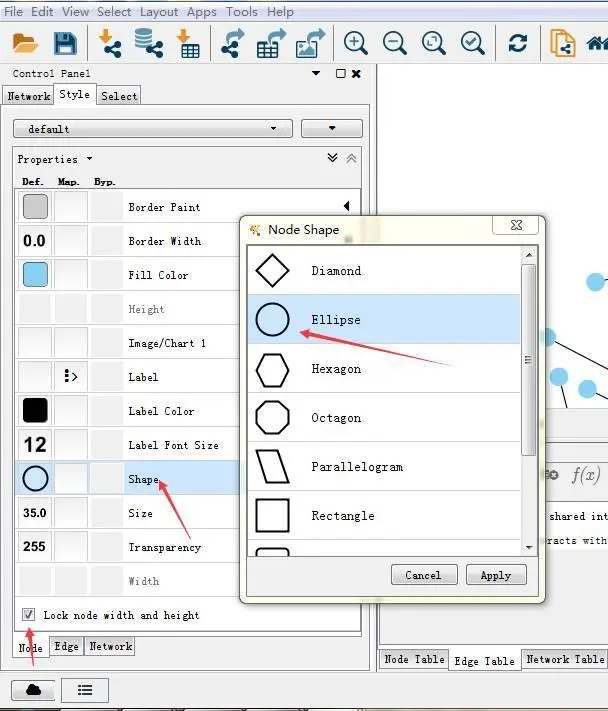
根据的molecluar type标注节点所代表lncRNA或者protein点击网络处理面板中style->node->shape进行设置点的形状,并勾选“Lock node width and height”。
点击网络处理面板中style->node->fill color进行设置点的颜色Column选择“molecluar type”和MApping Type选择“Discrete MApping”,并将lncRNA与protein设置成两种颜色区分

根据节点的大小来显示dregee大小,dregee值越大节点直径越大。

同样可以对点进行类似的细化设置(根据下列参数进行设置)

根据correlation的值调整边的颜色:点击网络处理面板中style->edge->stroke color进行设置Column设置为correlation,mApping type设置为continues mApping,current mApping(默认为白-黑)

注:可以双击灰色图标进行设置其它颜色,点击set min and max 设置数值,双击图形右上角的倒三角选择最大值显示的颜色;同样双击左上角倒三角选择最小值的颜色;如果需要增加图注,可点击“Add”按钮


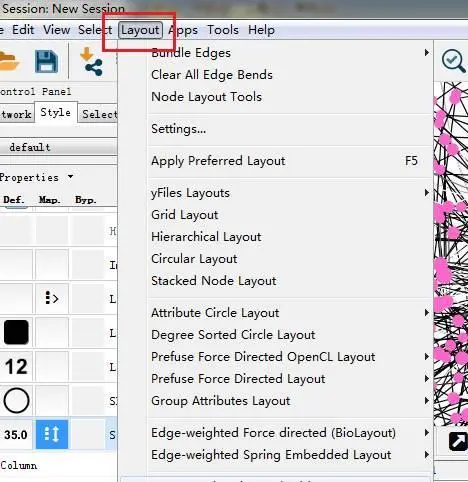
同样也可以对边进行细化设置最后再调整“Layout”得到满意的图
得到网络图:
最后点击菜单栏的图片导出*.pdf(同样可以采用export导出其它格式)注:注意调整网络图后再保存,否则会出现网络图不完整
四、子网提取
导入文件:file->import->network->file(ppi.txt)(HPRD中处理后的文件)
注:注意第一行是否为标题行,若是记得在advanced中去掉√节点选择:Select->node注:节点的选择可以通过将目标节点放在一个文件中导入,如果节点数少的话可以手动点选
注:黄色点为所选的目标节点
若想把其与大网络分离的话就点击菜单的图标栏里倒数第四个图标,获得子图
同样可对子网进行修饰如:改变节点颜色性质并将蛋白质换成对应的gene symbol(label中设置)
最后导出图片用MCODE插件,筛选出子网注:Mcode下载地址:http://Apps.cytoscape.org/Apps/mcode导入文件:file->import->network->file(net.txt)用MCODE插件筛选子网,参数可根据数据自行设置
分析后得到5个子网
可以将它创建到主窗口进行style调整或者导出创建到主窗口后
对创建到主图的子图进行修饰
保存子网即可MCODE详细介绍可参考http://baderlab.org/Software/MCODE/UsersManual
四、String-蛋白质相互作用数据库
STRING是蛋白质相互作用数据库,可进行是搜索已知蛋白之间和预测蛋白质之间相互作用主页:
按照蛋白质名称,氨基酸序列等信息进行检索某个特定的蛋白质相互作用的其他蛋白质例如:TP53 人类,选择人类
继续点continue,得到检索结果
圆圈代表蛋白质,点击圆圈可以查看蛋白质相关信息

直线代表蛋白质之间相互作用,点击可以查看互作信息

Setting中可以设置网络边代表的意义evidence:不同颜色的线表示不同证据 confidence:两个蛋白质相互作用越强连线越粗 actions:不同颜色和形状的线表示不同的作用

例如confidence

cluster聚类

得到结果

还可以对网络进行分析、视图放大缩小或保存输入某些蛋白质名称或氨基酸序列,检索其相互作用关系

例如:输入TP53、BCL2、MDM2、CDK2,并选择人类检索结果

String与Cytoscape联用
将String的蛋白互作数据下载到Cytoscape本地中Cytoscape中Apps->stringApp

安装完毕后,回到文件>import>network>public databases选择string数据库,以基因或蛋白输入

以TP53为例,选择score为0.2,连接的基因最大为80个。

也可以在已构建好的网络中可以扩展新的连接蛋白数或者重设confidence score来调整网络大小。

例如重新调整confidence score


可以进一步保存修改调整网络

