尔云间生信代码|R的Pathifier包获取疾病异常通路
普通的差异表达分析已经不能满足生物信息工作者对表达谱数据的分析要求,生信工作者希望能够使用更加全面,更加有说服力的方法来分析表达谱数据。在此我就向大家介绍一个主要针对于癌症的样本基因表达谱进行通路失常得分的R包。
科研有捷径,输入代码,一键获取科研成果!就是这么省事,来具体看下有多方便!
搜索http://985.so/a9kb查看全部代码(目前共计50+持续新增中),也可以点击右侧【目录】,可以看到更多有趣的代码;真香提示:文末可以知道如何获取代码~
随着生物数据的获得越来越快,特别是基因的表达谱数据每天都以大数量级的数量在增长,而对于基因表达谱的分析方法也越来越渴求。
普通的差异表达分析已经不能满足生物信息工作者对表达谱数据的分析要求,生信工作者希望能够使用更加全面,更加有说服力的方法来分析表达谱数据。
在此我就向大家介绍一个主要针对于癌症的样本基因表达谱进行通路失常得分的R包。
这个失常得分主要是通过癌症样本与正常样本在通路上的拟合距离所计算得来的,如果想要了解具体的计算原理。
可以看(Drier Y, Sheffer M, Domany E. Pathway-based personalized analysis of cancer. Proceedings of the National Academy of Sciences, 2013, vol. 110(16) pp:6388-6393. (www.pnas.org/cgi/doi/10.1073/pnas.1219651110))这篇文献,下面我来介绍一下pathifier R包的具体使用方法。
在R中通过Bioconductor就可以获得该包。

他的使用命令行为:
quantify_pathways_deregulation(data, allgenes, syms, pathwaynames, normals = NULL,ranks = NULL, attempts = 100, maximize_stability = TRUE, logfile = "", samplings = NULL,min_exp = 4, min_std = 0.4)
下面我来介绍每个参数的意义,以及我们所需要准备的数据格式(数据我以从TCGA上下载的GBM的癌症表达谱数据,以及KEGG中的通路数据为例)。
data:是一个N*M的mRNA表达矩阵,其中N是基因的个数,M是样本的个数。部分样本格式如下图。

allgenes:是N个基因的名字,geneID或者geneSymbol都可以,但是要和下面的通路数据相对应。如下图所示:

Syms:是P个通路的list,每个通路都是它包含的基因list(这个名字必须和上述的allgenes用相同的表示方式)。如下图所示。
pathwaynames:通路的名称,可以是hsaID或者直接是通路的名字,为了计算简单,只以两个通路为例。
normals :一个表示样本是否为正常样本的list,例如我们的数据最后十列为正常样本,其他均为癌症样本,则这个list只有最后十个为TURE,其余均为FALSE。
ranks :如果要把M个样本重新排序需要用到这个参数,一般都不会用到。
attempts :规定重复多少遍来测试数据的可靠性,一般情况下都设为100.
maximize_stability:如果这个设为TRUE,则会将程序认为会使得样本低稳定性的样本去掉。
logfile :LOG文件输出名字,可省略,省略则用默认的log名.
samplings :规定重新选择样本,默认则随机选择。
min_exp :最小的表达值
min_std :每个基因的表达值所允许的最低标准差,如果基因的标准差低于这个阈值,则程序会用算法标准化其标准差。
我将我所使用的命令行贴上

而我所使用的测试数据也一并上传,分别是test.txt(表达谱数据),geneList.txt(基因名),normal.txt(哪些是正常样本)。(数据地址:http://pan.baidu.com/s/1dDkvPwH)
经过一段时间的计算我们可以获得一个PDS(通路失常得分)。而这个PDS得分也是以list形式给出,下面是PDS这个list里各个组成部分的内容。
scores :失常得分,也就是pathifier最主要的结果。
genesinpathway :在计算失常得分时每个通路中使用到的基因。
newmeanstd :去除噪声点后,重新计算获得的标准差。
origmeanstd :去除噪声点前,计算获得的标准差。
pathwaysize :计算通路得分所使用基因的数目。
curves :每个通路的PC(文献中有介绍)。
curves_order :PC顺序。
z :在计算PC的时候的Z-scores
compin :因为噪声所去除的组分(通路中的基因)
xm :正常样本均值
xs :正常样本标准差
center : PCA的中心
pctaken :主成份的数目
sucess :成功计算了PDS的通路
logfile :LOG文件的名称(之前没有规定则为默认名字)
下面我主要展示一下我所用包中的实例数据所做出的主要score结果。(我所上传的数据主要是为了方便大家将类似的数据通过我上传的语句尽快处理成R包所需格式,方便计算)

本实例中一共涉及两个通路,也即是MISMATCH_REPAIR和REGULATION_OF_AUTOPHAGY,这就是样本对于这个通路的失常得分。
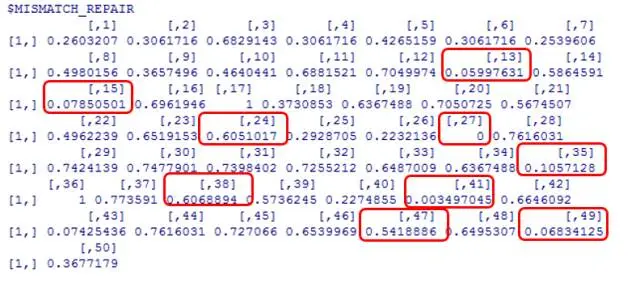
上图中红色圈出来的就是实例样本中的正常样本,可以发现,正常样本在通路上的失常得分普遍较低,说明在该通路上并未失常。
大家学会这个R包后,就能很方便的通过通路来分析不同样本的失常得分。或者可以通过自己所感兴趣的基因集也能得到类似的结果。
查看原文,请扫码


