【爆肝万字】基于Rosetta的配体结合位点的设计(Rosetta,Design of Ligand Binding)
1. 前言
Rosetta是蛋白质设计中的重要平台,在蛋白质设计领域中占有重要地位。本文以2016年的文献Rosetta and the Design of Ligand Binding Sites进行实践,探索用Rosetta进行配体结合位点的设计。经过三个月实践成功,得到本文档,在此分享给读者,希望能够帮到有需要的读者完成实验工作,早发文章!
觉得有用还请点个赞呀!ヾ(◍°∇°◍)ノ゙
文献信息:Methods Mol Biol. 2016 ; 1414: 47–62. doi:10.1007/978-1-4939-3569-7_4.
Rosetta and the Design of Ligand Binding Sites - PubMed

总体设计流程如下图所示。
2. 材料
(1)运行类Unix操作系统(如Linux或MacOS)的计算机。尽管测试运行和小型生产运行可以在传统的笔记本电脑和台式机系统上执行,但建议在生产运行中使用多处理器计算群集。
(2)Rosetta。Rosetta建模包可以从RosettaCommons网站(https://www.rosettacommons.org/software/license-and-download)获得。Rosetta许可证免费提供给学术用户。Rosetta以源代码形式提供,必须在使用前编译。有关如何编译Rosetta的说明,请参阅Rosetta文档(https://www.rosettacommons.org/docs/latest/)。本文中的方案已使用Rosetta每周发布版本2021.16.61629进行了测试。
特别注意:版本不同,使用时会有所差别,比如原文章中使用的是2016版本的Rosetta,具体到2021年的版本是有些差异的。
(3)处理小分子的程序。OpenBabel是一个自由软件包,允许操作许多小分子文件格式。有关下载和安装信息,请访问http://openbabel.org获取下载和安装信息。本文中的协议已在OpenBabel版本2.3.1中进行了测试。也可以使用其它小分子操作程序。
(4)配体构象生成程序。我们推荐BCL,可从http://meilerlab.org/index.php/bclcommons免费获得,用于学术用途,但需要剑桥结构数据库的额外许可证才能生成构象异构体。本文中的协议已使用BCL 3.2版进行了测试。也可以使用其他构象异构体生成程序,如Omega、MOE或RDKit。
(5)标准格式的靶小分子结构,如SDF或SMILES。
虽然Rosetta可以忽略远离结合位点的链断裂和缺失环,但蛋白质的结构在配体结合区域应该是完整的。如果结合口袋缺失残基,使用起始结构作为模板,用比较建模方案重塑这些残基。
(6)待重新设计的蛋白质结构,PDB格式。
可接受的格式取决于小分子处理程序的能力。OpenBabel可用于将大多数小分子表示(包括SMILES和InChI)转换为Rosetta所需的sdf格式。
在与密切相关的配体的络合物中测定的高分辨率实验结构是最理想的,但不是必需的。可以使用未配体化蛋白质的实验结构甚至同源性模型。
3. 方法
总述:在整个方案中,${ROSETTA}表示安装Rosetta的目录。文件内容和要在终端中运行的命令以斜体显示。假定使用bash shell-其他shell的用户可能需要修改命令行的语法。
参照本部分理论,使用Rosetta的relax protocol优化蛋白结构,在存放蛋白结构文件(以DEBP.pdb为例)的目录下打开Linux命令行界面。我本地Rosetta安装路径为:
/home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle
注意:运行时需要找到个人安装Rosetta的路径进行替换。
3.1 将蛋白质结构预松弛到Rosetta评分函数
具体命令如下:
/home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/bin/relax.mpi.linuxgccrelease -ignore_unrecognized_res -ignore_zero_occupancy false -use_input_sc -flip_HNQ -no_optH false -relax:constrain_relax_to_start_coords -relax:coord_constrain_sidechains -relax:ramp_constraints false -s DEBP.pdb
命令正确,开始运行,输出日志!

运行结束后输出文件为DEBP_0001.pdb。

为方便起见,重命名输出结构。可以直接在xftp中更改文件名,或用mv命令更改文件名(命令如下)。
mv DEBP_0001.pdb DEBP_relaxed.pdb
3.2 准备配体
3.2.1 将小分子转化为SDF格式,包括根据需要添加氢
从RCSB数据库中下载AKG的sdf小分子构象文件AKG_ideal.sdf。
使用OpenBabel软件为AKG补加氢原子,在存放配体构象文件的目录下打开Win10命令行窗口。

输入以下命令:
obabel AKG_ideal.sdf -p 7.4 -O AKG.sdf
其中,-p指定在pH=7.4的条件下为AKG加氢。程序运行结束后会输出氢原子数符合体液pH条件的AKG构象AKG.sdf。
运行后显示“1 molecule converted”,即一个分子已经转化完成。

在原路径下新生成了AKG.sdf这个文件。

以下对OpenBabel软件处理前后的sdf文件做一下简单对比,可以发现处理后去掉了两个氢原子。即程序认为4个氢原子符合体液pH为7.4的条件。
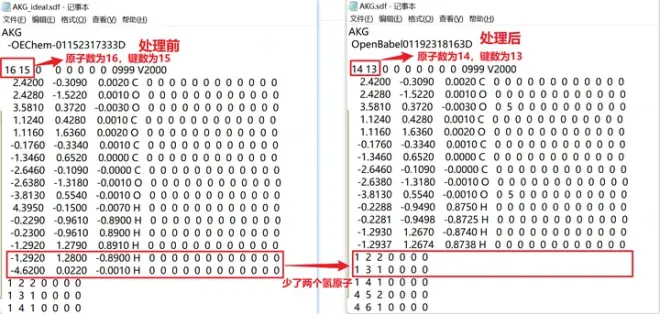
分析AKG的结构可以发现一些端倪,AKG中含有两个羧基,在ph设置为7.4的弱碱性环境下,可以猜想去掉两个氢原子是合理的。

后续尝试将PH设置为14,强碱性环境下失去两个氢原子,而PH=1的强酸性环境下没有去掉氢原子,即酸性环境下AKG不会失去氢原子,这和之前的猜想是一致的。

3.2.2 生成配体构象异构体库,将构象异构体库转换为Rosetta格式的“参数文件”
不同于蛋白大分子,配体小分子一般都具有相当大的灵活性,同一分子会以不同的构象与蛋白发生相互作用。因此,为了在设计时考虑到小分子的灵活性,需要生成小分子的构象异构体集合。
原文献中使用的是BCL的本地应用程序,而以下使用更为简便的BCL::Conf网络服务器生成配体构象异构体库。
从[文献](https://pubmed.ncbi.nlm.nih.gov/33351632/)中了解到BCL下载,以及BCL::Conf网络服务器的网址(如下),二者均可以实现生成配体构象异构体库,但实际使用时打不开。
BCL下载:http://meilerlab.org/bclcommons
BCL::Conf网络服务器:http://meilerlab.org/bclconf
之后通过检索找到可用的[BCL::Conf网络服务器](Meiler Lab)。

在 Molecules选项中上传已加氢的AKG.sdf;在Conformer Generation Mode选项中选择Standard;在Number of Conformers Desired / Molecule选项中填写100。

提交设置和任务后,提示在页面要等待大约48秒。
结果不到30秒就自动刷新页面显示了结果。

点击conformers_bcl_conf.sdf文件即可下载,该文件中含有100个加氢后的AKG分子的构象。将其重命名为AKG_conf.sdf以便进行后续操作。
接下来将该构象集合转换为 Rosetta 可读取的、用于储存小分子形状及化学性质信息的专有格式:params 文件。在存放构象异构体集合文件(AKG_conf.sdf)的目录下打开 Linux 命令行界面,输入以下命令:
${ROSETTA}/main/source/scripts/python/public/molfile_to_params.py -n LIG -p LIG -- conformers-in-one-file AKG_conf.sdf
其中,-n 指定在 pdb 和 params 文件中用来表示配体名称的 3 字符缩写,这里命名为 LIG 即配体 ligand 的缩写(需要注意的是,这里不能沿用 GLY 或者其他氨基酸、金属原子的缩写,否则生成的 params 文件会在后续突变结构时,与 Rosetta 自带的氨基酸或部分金属原子的 params 文件发生冲突);-p 指定生成文件的命名。
我实际输入命令(${ROSETTA}代表的路径不同,需替换成本地的路径):
/home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/scripts/python/public/molfile_to_params.py -n LIG -p LIG --conformers-in-one-file AKG_conf.sdf
输出正常,很快运行完成。
在Xftp中查看输出文件,可以看到新产生了3个文件:配体形状及化学信息文件 LIG.params,Rosetta 选定的 1 个配体构象LIG.pdb,配体构象集合 LIG_conformers.pdb。

用PyMOL查看LIG.pdb和LIG_conformers.pdb的结构,分别如下图所示。


至此,配体准备工作完成。
3.3 将配体置于蛋白质中
Rosetta要求原子名称与molfile_to_params.py步骤中生成的名称匹配。即使具有正确放置了配体的起始结构,也应该将molfile_to_params.py生成的结构对齐到口袋中,以便原子命名正确。
在进行蛋白设计时会包含蛋白与配体的多次对接,因此需要先指定配体每个原子的起始坐标,这样配体才能在指定的对接盒子中移动和扭转,与结合口袋发生相互作用。
为此,只需要在PyMOL中打开蛋白结构、以及前文Rosetta选定的其中1个配体构象LIG.pdb,将配体移动至结合口袋内即可。
为了提高定位的准确性、缩小对接需要搜索的空间范围,在PyMOL中打开含原配体Glu 的蛋白结构2VHA.pdb和 LIG.pdb,将鼠标操作改为编辑模式,移动和旋转LIG.pdb,使之与2VHA.pdb中的原配体Glu在羧基处大致重合,如下图所示。

调整好LIG.pdb的位置之后,删除原配体Glu和蛋白DEBP的复合物结构2VHA.pdb,只留下已移动到结合口袋处的AKG,保存为 LIG_positioned.pdb。
这个过程相对简单,实际操作中需要注意一些细节,以下进行简单说明。
(1)如何在PyMOL中设置鼠标操作为编辑模式?
在界面右下角可以看到“Mouse Mode”,即鼠标操作模式。
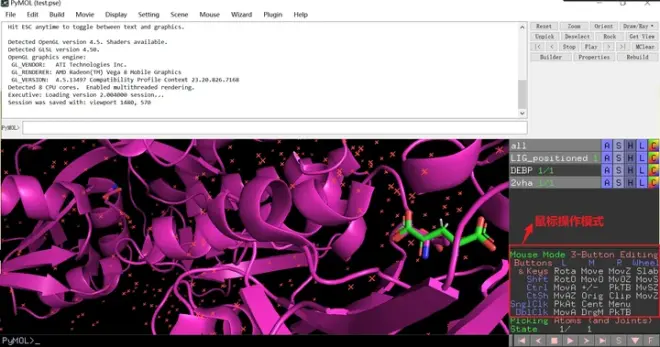
只需在上图红框区域单击鼠标即可实现显示模式(3-Button Viewing)和编辑模式(3-Button Editing)的切换。
(2)在PyMOL中调整配体位置过程中的细节
因为上述操作过程中,我们只想移动配体LIG.pdb,而不想蛋白质2VHA.pdb位置发生改变,因此可以在显示模式对蛋白进行固定,固定方式为:选中蛋白结构→A(ACTION)→MOVEMENT→PROTECT。
在编辑模式下,一些鼠标操作如下:
Shift键+鼠标左键——旋转配体;
Shift键+鼠标中键——可平移配体;
长按鼠标右键滑动——缩放视野;
Ctrl+Shift+鼠标中键——将鼠标所在位置设置为旋转中心。
(3)为蛋白质设置透明度和透明方式
在移动和旋转配体时,不透明的蛋白显示方式会造成遮挡,不便于操作。因此可以在显示模式下选中蛋白质,为其设置透明度,如果显示的方式为cartoon,将其设置成透明度设置为50%,可以按以下步骤操作:点击菜单栏中的Setting→Transparency→cartoon→50%。
更多透明度和透明方式的设置,可参见[PyMOL中文教程](入门教程 - PyMOL中文教程 2022.09 文档)。
(4)检查
调整好LIG.pdb的位置,保存为LIG_positioned.pdb之后,可以重新打开LIG_positioned.pdb和2VHA.pdb查看重合情况。如下图可以看到,LIG_positioned.pdb和原配体Glu重合情况较好。

3.4 运行Rosetta设计
3.4.1 突变位点声明
实际操作时,这个步骤很简单,就是新建一个.txt文档,在其中输入以下文字,保存后将文档后缀名由`.txt`改为`.resfile`即可。
ALLAA AUTO start 1 × NATAA
说明:
其中,“start”以上为 Header 部分,“start”以下为 Body 部分。
Header 声明了对所有未在 Body 中声明的残基要采取何种操作:“ALLAA”即允许选定残基突变为 20 种常规氨基酸中任意一种;“AUTO”即让 Rosetta 自动检测结合界面确定待突变残基。即 Header 允许位于结合界面的残基突变为 20 种常规氨基酸中任意一种,其他位置的残基不发生突变。
Body 声明的操作作为 Header 的例外被优先确定,即当 Header 和 Body 对同一残基采取的操作发生冲突时,以 Body 为准。“1 X NATAA”即允许 X 链第 1 位残基采取 repack操作,即发生构象变化。但 1R9L 的蛋白结构只有 A 链,关于 X 链的情况需要特别说明:在 3.2.3 配体文件准备与预处理中,将构象集合 sdf 文件转换为 params 文件时,生成的LIG.pdb,LIG_conformers.pdb 都会将 GLY 放置在 X 链;LIG.params 中 MM AtomType项也设置为 X。这是因为 Rosetta 默认 X 链是配体、配体原子类型是 X。因此 X 链第 1位残基就是指目标配体 GLY。即 Body 只允许配体构象变化,不允许配体发生突变。
附:浅谈Rosetta Mutfile与Resfile用法 - 知乎
谷雨:浅谈Rosetta Mutfile与Resfile用法
补充:Commands for for controlling sequence identity(控制序列识别的命令)
参考自(下面是对内容的翻译):Resfile syntax and conventions
每个命令都用于限制每个位置允许的氨基酸种类。如果组合多个命令,只有被每个命令单独允许的氨基酸才会被包含。这是由于对PackerTask类操作的交换性质。
注意:应该记住,resfile命令是限制性的,而不是允许性的。虽然不允许的残基不会被重新打包/重新设计,但是各种协议提供了额外的限制,可能进一步限制突变身份。特别地,许多协议会禁止重新设计二硫键半胱氨酸,即使在resfile中明确列出为可突变。
每个命令都用于限制每个位置允许的氨基酸种类。如果组合多个命令,只有被每个命令单独允许的氨基酸才会被包含。这是由于对PackerTask类操作的交换性质。 注意:应该记住,resfile命令是限制性的,而不是允许性的。虽然不允许的残基不会被重新打包/重新设计,但是各种协议提供了额外的限制,可能进一步限制突变身份。特别地,许多协议会禁止重新设计二硫键半胱氨酸,即使在resfile中明确列出为可突变。
ALLAA … 允许所有20种氨基酸,包括半胱氨酸(与ALLAAwc相同)
ALLAAwc … 允许所有20种氨基酸(默认)
ALLAAxc … 允许除半胱氨酸外的所有氨基酸
POLAR … 只允许极性的标准氨基酸(DEHKNQRST)
APOLAR … 只允许非极性的标准氨基酸(ACFGILMPVWY)
PROPERTY <property> … 不允许任何缺乏给定属性的残基类型
NOTAA <list of AAs> … 只不允许指定的氨基酸(使用单字母代码,不用分隔符,如ACFYRT。对于非标准氨基酸,使用X[<full name>]。)
PIKAA <list of AAs> … 只允许指定的氨基酸(使用单字母代码,不用分隔符,如ACFYRT。对于非标准氨基酸,使用X[<full name>]。)
NATAA … 只允许本来的氨基酸(NATive Amino Acid)- 重新打包而不设计
NATRO … 保留输入的旋转异构体(完全不打包)(NATive ROtamer)
3.4.2 设计protocol
准备对接和设计脚本(“design.xml”)。建议的方案基于使用RosettaScripts框架的RosettaLigand对接。它将优化配体在结合口袋中的位置(low_res_dock),重新设计周围的侧链(design_interface),并在设计的环境中优化相互作用(high_res_dock)。为了避免虚假突变,在每个位置(favor_native)给输入残基一个轻微的能量奖励。
新建一个记事本文件,将以下内容写入,并将文件另存为design.xml即可。
<ROSETTASCRIPTS> <SCOREFXNS> <ScoreFunction name="ligand_soft_rep" weights="ligand_soft_rep.wts"/> <ScoreFunction name="hard_rep" weights="ligandprime.wts"/> </SCOREFXNS> <TASKOPERATIONS> <DetectProteinLigandInterface name="design_interface" cut1="6.0" cut2="8.0" cut3="10.0" cut4="12.0" design="1" resfile="DEBP.resfile"/> </TASKOPERATIONS> <LIGAND_AREAS> <LigandArea name="docking_sidechain" chain="X" cutoff="6.0" add_nbr_radius="true" all_atom_mode="true" minimize_ligand="10"/> <LigandArea name="final_sidechain" chain="X" cutoff="6.0" add_nbr_radius="true" all_atom_mode="true"/> <LigandArea name="final_backbone" chain="X" cutoff="7.0" add_nbr_radius="false" all_atom_mode="true" Calpha_restraints="0.3"/> </LIGAND_AREAS> <INTERFACE_BUILDERS> <InterfaceBuilder name="side_chain_for_docking" ligand_areas="docking_sidechain"/> <InterfaceBuilder name="side_chain_for_final" ligand_areas="final_sidechain"/> <InterfaceBuilder name="backbone" ligand_areas="final_backbone" extension_window="3"/> </INTERFACE_BUILDERS> <MOVEMAP_BUILDERS> <MoveMapBuilder name="docking" sc_interface="side_chain_for_docking" minimize_water="true"/> <MoveMapBuilder name="final" sc_interface="side_chain_for_final" bb_interface="backbone" minimize_water="true"/> </MOVEMAP_BUILDERS> <SCORINGGRIDS ligand_chain="X" width="15"> <ClassicGrid grid_name="vdw" weight="1.0"/> </SCORINGGRIDS> <MOVERS> <FavorNativeResidue name="favor_native" bonus="1.00"/> <Transform name="transform" chain="X" box_size="3.0" move_distance="0.1" angle="5" cycles="500" repeats="1" temperature="5" rmsd="4.0"/> <HighResDocker name="high_res_docker" cycles="6" repack_every_Nth="3" scorefxn="ligand_soft_rep" movemap_builder="docking"/> <PackRotamersMover name="design_interface" scorefxn="hard_rep" task_operations="design_interface"/> <FinalMinimizer name="final" scorefxn="hard_rep" movemap_builder="final"/> <InterfaceScoreCalculator name="add_scores" chains="X" scorefxn="hard_rep"/> <ParsedProtocol name="low_res_dock"> <Add mover_name="transform"/> </ParsedProtocol> <ParsedProtocol name="high_res_dock"> <Add mover_name="high_res_docker"/> <Add mover_name="final"/> </ParsedProtocol> </MOVERS> <PROTOCOLS> <Add mover_name="favor_native"/> <Add mover_name="low_res_dock"/> <Add mover_name="design_interface"/> <Add mover_name="high_res_dock"/> <Add mover_name="add_scores"/> </PROTOCOLS></ROSETTASCRIPTS>
注:在XML脚本中更改resfile的名称,使其与正在使用的resfile的完整路径和文件名相匹配。切割值决定如何使用AUTO规格处理残留物。将设计在配体的切口1埃内具有C-β原子的所有AUTO残基,以及切口2内指向配体的所有残基。选择侧链的逻辑分别类似于切口3和切口4,但具有侧链灵活性而不是设计。在设计阶段将忽略剖切壳之外的任何内容,但在其他阶段可能会移动这些内容。
注:网格宽度必须足够大以容纳配体。对于较长的配体,请将该值至少增加到配体的最大延伸长度加上“变换”移动器中box_size值的两倍。
注:box_size的值设定配体从起始位置的最大刚体置换。rmsd的值设置与起始位置的最大允许均方根偏差。如果希望使设计的配体更接近起始构象,请将这些值设置为较小的值;如果希望允许更多的移动,请将这些值设置为较大的值。这些仅是方案主动采样阶段的限值。在协议的其他阶段可能会发生额外的移动。
3.4.3 运行设计应用程序
输入以下指令运行。-nstruct 5000代表生成5000个pdb文件,可根据需要进行更改。
/home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/bin/rosetta_scripts.mpi.linuxgccrelease -ex1 -ex2 -linmem_ig 10 -restore_pre_talaris_2013_behavior -parser:protocol design.xml -extra_res_fa LIG.params -s "DEBP_relaxed.pdb LIG_positioned.pdb" -nstruct 5000 -out:file:scorefile design_results.sc
在目录下生成了所需的pdb文件。

3.5 进行两轮筛选
(1)第1次筛选
为了查看高分结构,在当前目录下打开 Linux 命令行界面,输入以下命令:
awk '{print $NF,$2}' design_results.sc | sort -nk2 > sorted_scores.txt
程序运行结束后会输出20,000个结构按total_score项排列的分数记录文件,该项代表总体得分的大小,一般为负数,负得越多说明能量越小,结构越稳定。
为了获得上述各项的平均值,需要先过滤一次以查看附带输出的各项平均值信息。创建之后要写入过滤参数的空文件metric_thresholds.txt,在当前目录下打开 Linux 命令行界面,实际输入以下命令:
perl /home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/src/apps/public/enzdes/DesignSelect.pl -d <(grep SCORE design_results.sc) -c metric_thresholds.txt -tag_column last > filtered_designs.sc
命令行界面在程序一开始执行时会输出各项得分的平均值,将过滤时要用到的得分项平均值记下,删除本次输出的 filtered_designs.sc(也可不删除,因下次过滤输出的结果会覆盖此文件)。

根据过滤项平均值,打开metric_thresholds.txt写入过滤参数。写入内容如下:
req total_score value < -1238.12 req fa_rep value < 97.62 req if_X_fa_atr value < -12.93 req if_X_fa_rep value < 1.92 req ligand_is_touching_X value > 0.8 output sortmin total_score
在当前目录下打开Linux命令行界面,再次输入上次过滤的命令,程序运行结束后会输出filtered_designs.sc文件,文件包含了按interface_delta_X项排序的滤过结构列表。

(2)第2次筛选
进入到要产生构象的文件目录下,输入以下命令:
awk '{print $NF ".pdb"}' filtered_designs.sc >filtered_pdbs.txt
Rosetta的InterfaceAnalyzer计算了许多额外的指标。不过,这些可能需要时间来评估,因此最好只在一组经过预过滤的结构上运行。在生成指标之后,可以如前面的步骤中那样过滤结构。这将生成一个分数文件(“design_interfaces.sc”),其中包含所选PDB的计算的指标值。
实际使用指令:
/home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/bin/InterfaceAnalyzer.mpi.linuxgccrelease -interface AB_X -compute_packstat -pack_separated -score:weights ligandprime -no_nstruct_label -out:file:score_only interfaces.sc -l filtered_pdbs.txt -extra_res_fa LIG.params
这步比较费时,相当于在第一步筛选结果的基础上,进一步计算新的指标并进行筛选,相当于计算一千多个构象的新的指标。运行结束之后得到interfaces.sc文件,包含第1次筛选后留下的1168个构象。
为了获得上述各项的平均值,需要先过滤一次以查看附带输出的各项平均值信息。创建之后要写入过滤参数的空文件metric_thresholds_2.txt,在当前目录下打开 Linux 命令行界面,输入以下命令:
perl /home/hdeng/software/rosetta_bin_linux_2021.16.61629_bundle/main/source/src/apps/public/enzdes/DesignSelect.pl -d <(grep SCORE interfaces.sc) -c metric_thresholds_2.txt -tag_column last > filtered_interfaces.sc
运行后上述命令之后各项指标的平均值输出如下。

根据过滤项平均值,打开metric_thresholds_2.txt写入过滤参数。写入内容如下:
req packstat value > 0.75 req sc_value value > 0.75 req delta_unsatHbonds value < 4.82 req dG_separated/dSASAx100 value < -0.81 output sortmin dG_separated
在当前目录下打开Linux命令行界面,再次输入第2次过滤的命令,程序运行结束后会输出filtered_interfaces.sc文件,文件包含了按dG_separated项排序的滤过结构列表和各项分数,总共筛选得到93个构象。

通过运行以下命令可以得到只包含已通过筛选的文件的名称的文件filtered_pdbs_final.txt。
awk '{print $NF ".pdb"}' filtered_interfaces.sc >filtered_pdbs_final.txt

至此,通过Rosetta进行筛选的步骤结束。
3.6 手动检查选定序列
虽然自动化程序在不断改进,并且可以在有限程度上替代,但在评估设计时,仍然没有人的专业知识可以替代。领域专家对界面的视觉检查可以捕获难以编码到自动过滤器中的系统特定需求。
3.7 重新应用设计方案,从步骤3.4开始
通过对来自前几轮设计的输出结构重复设计协议,可以获得改进的结果。设计轮次的数量取决于系统及其收敛速度,但通常需要3-5轮设计,每轮设计都从上一轮的过滤结构开始。
在后续循环中,随着配体稳定到首选结合方向,可能希望降低低分辨率采样阶段的积极性(步骤3.4.2中“变换”移动器的box_ size和rmsd值)。由于输出结构同时包含蛋白质和配体,因此传递给“-s”选项的值上不再需要引号(参见步骤3.4.3)。与之相反,你可能希望使用“-l”选项,该选项采用每行包含一个输入PDB的文本文件的名称。每个输入PDB都将生成“-nstruct”模型。减小此值,使每轮中未筛选的输出结构的总数大致相同。
3.8 设计迭代
根据计算协议进行的初始设计很少能给予精确的预期结果。通常需要执行设计和实验的迭代循环,使用从实验中获得的信息来改变设计过程。

