科普硬货-靶向测序概述
自从1977年一代测序被发明以来,测序技术不断发展。 从一代sanger测序,发展到二代测序,现在已经到了三代全长测序。 其中二代测序相比于一代测序,通过将核酸片段化进行平行大规模测序,大大增加了测序的效率。早期的二代效率集中在全基因组测序和全转录组测序,尽管测序的效率有所增加,成本有所降低。 但是测序数据依然非常庞大,为了更进一步的节省成本,提高效率,靶向测序应运而生。
靶向测序定义:
将基因组中感兴趣的区域或者位点富集出来,然后使用二代测序(NGS)方法去进行测序,包含全外显子组(基因组蛋白编码区域),针对感兴趣的特定基因定制测序panel等。
靶向捕获测序背景
1977年 Walter Gilbert和Frederick Sanger发明了第一台测序仪,使用链终止法测序其测定了第一个基因组序列,噬菌体X174,全长5375个碱基。Sanger 测序的发明,标志着基因测序技术正式进入生命科学研究舞台 1988年Chambehian等人首次提出多重PCR技术,为后续多重PCR扩增子测序打下基础 2005年Nature Method 上发表了一篇名为《Direct genomic selection》的文章,该文章利用长度为150kb生物素标记的BAC DNA和经过处理的人类基因组DNA进行杂交,通过链霉亲和磁珠对DNA片段进行捕获,后续又经过PCR扩增后进行测序。测序结果表明约~50%的序列来自于靶标区域

2008年,安捷伦联合Broad研究所,将其超长寡核苷酸合成技术和平行测序相结合,在Nature Biotechnology发表文章,奠定安捷伦杂交捕获测序方法学基础。

2009年,安捷伦联合华盛顿大学在Nature上发表文章,使用靶向捕获测序技术检测人类外显子

同年安捷伦推出了
世界上第一款商品化人类全外显子探针产品
。 2021 年 4 月,安捷伦宣布首款基于机器学习探针设计方案的全外产品—人全外显子组 V8 (SureSelect Human All Exon V8) 正式在中国上市,继续书写人全外显子靶向捕获技术新篇章。 2022年春, Qiagen 基于多重PCR技术全新一代 QIAseq Targeted DNA Pro 在中国正式上市 目前常用的靶向测序用两种方法:
靶向捕获法和多重PCR法(又称扩增子测序)
杂交捕获法
杂交捕获法是一种把
分子杂交
和
二代测序
相结合的靶向测序技术。 该技术需要设计和生产和目的区域互补的探针,通过探针将目的区域的片段捕获下来,再将不需要的部分进行洗脱。 根据杂交的状态又可分为固相杂交和液相杂交。
固相杂交
就是将设计好的探针固相的芯片上探针,通过探针将目标区段捕获。
液相杂交
的实验反应是在液体状态中完整,探针携带生物素,当杂交完成后,通过链酶亲和磁珠将探针吸附下来(此时探针有携带目标区段的和空探针),未被捕获的片段被洗脱掉,再通过变性将探针和目标片段分开,然后利用磁珠将所有空探针吸附丢弃,完成捕获。

图 1 安捷伦杂交捕获测序流程
多重PCR法
多重PCR靶向测序技术又称
扩增子靶向测序技术
,是一种将
多重PCR技术
与
二代测序技术
相结合的一种靶向测序技术。 该技术首先利用多重PCR反应,同时扩增多个目标区域序列,得到扩增子产物,然后通过PCR反应或者酶连接反应,将二代测序所需的接头序列(adapter)引入到扩增子产物的两侧,得到扩增子文库,然后进行二代测序和生信流程分析,获取目标区域的序列信息,实现目标区域序列检测的目的。常见的多重PCR靶向测序举例:tNGS病原微生物靶向测序,用于分析病原微生物的群落组成和分布,来进行临床病原微生物的诊断。

图 2 Qiagen 基于SPE技术的多重PCR靶向测序流程
全基因组 vs 全外显子 vs 多重PCR
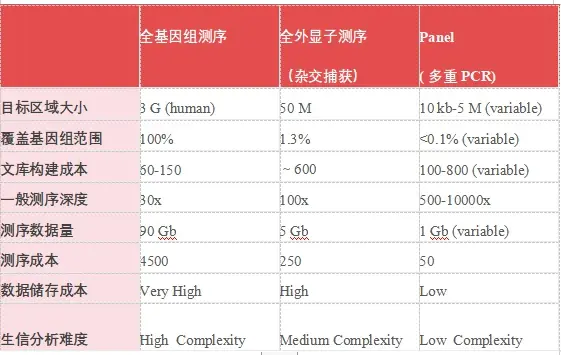
数据评估
目标基因区域捕获的数据质量主要通过以下指标评价:目标区域覆盖度、捕获效率、目标区域覆盖均一性等。
目标区域覆盖度:
指检测到的区域相比目标区域的比例,最理想的情况就是感兴趣的目标区域都能够被覆盖到。但是由于在设计探针的时候会考虑各种因素,如GC含量、序列的特征、序列的拷贝数,序列相似性等问题,为了保证整体的基因捕获效率,会选择放弃一小部分区域的捕获,这个比例约为0-3%。原则上来讲,目标覆盖度越高,探针或者多重PCR产品的性能也就越好。
捕获效率:
落在目标区域的数据占总数据的比例。捕获效率越高,代表测序数据的利用率越高。另外在设计探针时,需要评估覆盖位置的序列特征,如果探针有很多落在重复序列区域,或者高拷贝序列区,则探针会结合较多的非目标区域。设计更加特异性的探针能够有效减少非特异序列的结合,提升捕获效率。 通常影响捕获效率的因素有以下几点: 1. 高GC区域 - UTRs 和 启动子区域通常是非常典型的高CG含量区域,这部分区域往往是低捕获效率,并且会增加这些区域和其他区域的捕获差别 2. DNA 质量- 投入的DNA质量较差, 例如FFPE样本提取的DNA,会产生捕获偏差,因为这样样本中部分区域往往比其他区域碎片更多。如果捕获不平衡,就会在下游 SNP s和其他形式的分析中产生偏差。建议用安捷伦自动化电泳仪器对核酸样本进行质控,例如2100生物分析仪,Tapestation分析仪,Fragment analyzer等 3. DNA 投入量 - Low input DNA 在建库过程中往往需要更多的PCR循环数来或足够量的预文库。增加PCR循环数,会造成更多的PCR duplicates, 会降低最终数据的有用信息。随着技术发展,目前靶向测序所需DNA投入量已由传统的微克级别下降至ng级别 4. Pseudogenes -会降低覆盖率的均匀性 5. DNA片段大小 - 建议片段大小应和探针设计大小想匹配以获得更大的捕获效率,建议用安捷伦自动化电泳仪器对样本核酸片段进行检测,例如2100生物分析仪,Tapestation分析仪,Fragment analyzer等 6. Repeat elements - 会降低reads在外显子组中分布的均匀性,导致需要更多的测序来检测新的SNP。
覆盖均一性:
指每个区域的覆盖深度是不是均匀。要想获得高均一性覆盖度的数据,在预文库构建时,要保证文库的均一性要好。例如文库构建时,采用无序列偏差的DNA片段化方法;采用对GC含量偏好性低的扩增酶;减少PCR富集的循环数;如果使用探针杂交捕获方法,探针设计时要更好的计算探针的结合能力,合理调整探针比例,实验过程中采用高度优化的杂交缓冲液进行捕获实验。 更多精彩,请查看公众号“优宁维分子生物学”,有超多文献解读与实验技术科普文章等你来看

优宁维分子生物学

