![]()

 我们的研究设计流程是为了发现免疫相关的特征 lncRNA 模型。TSCC:舌鳞状细胞癌。TCGA:癌症基因组图谱。从 TCGA 数据库中提取 TSCC 患者的 lncRNA 谱数据,并鉴定出 TSCC 和正常对照之间存在显着差异水平的 lncRNA。随后,进行 Pearson 相关分析,以确定差异表达的免疫相关 lncRNA。接下来,具有有意义的生存数据的 TSCC 患者(n= 145 名(排除 4 名缺少生存数据的 TSCC 受试者)被随机分为训练队列和测试队列(训练与测试队列比例:~2:1)。进行单变量和多变量 cox 回归分析来构建特征 lncRNA 模型。最后,使用测试队列来验证我们开发的lncRNA模型的功效,并进行后续的预后分析、ROC分析和PCA来评估特征lncRNA模型。
我们的研究设计流程是为了发现免疫相关的特征 lncRNA 模型。TSCC:舌鳞状细胞癌。TCGA:癌症基因组图谱。从 TCGA 数据库中提取 TSCC 患者的 lncRNA 谱数据,并鉴定出 TSCC 和正常对照之间存在显着差异水平的 lncRNA。随后,进行 Pearson 相关分析,以确定差异表达的免疫相关 lncRNA。接下来,具有有意义的生存数据的 TSCC 患者(n= 145 名(排除 4 名缺少生存数据的 TSCC 受试者)被随机分为训练队列和测试队列(训练与测试队列比例:~2:1)。进行单变量和多变量 cox 回归分析来构建特征 lncRNA 模型。最后,使用测试队列来验证我们开发的lncRNA模型的功效,并进行后续的预后分析、ROC分析和PCA来评估特征lncRNA模型。 结果
数据处理和差异表达分析
149 名 TSCC 受试者的临床病理学和转录组数据最初从 TCGA-HNSC 项目下载。从完整的转录组谱中总共提取了16,902个lncRNA,通过edgeR包进行差异表达分析,发现1108个lncRNA在TSCC和正常组织之间存在显着差异表达(调整p < 0.05和|log2FC| ≥ 1) 。TSCC 中显着变化的 lncRNA 的热图和火山图。利用ImmPort中的免疫相关基因列表(n = 2483),从TSCC数据集中提取1502个免疫相关基因的表达数据,然后通过Pearson相关分析1108个lncRNA与1502个免疫相关基因之间的表达量基因(mRNA),获得了 695 个差异表达的免疫相关 lncRNA(p < 0.001 和 |R| > 0.4)。这些免疫相关lncRNA对免疫相关基因的表达有正向或负向调节。
 TSCC ( n = 149) 和正常 ( n = 15) 组织样本之间差异表达 lncRNA 的热图 ( A ) 和火山图 ( B )。热图表明 TSCC 和正常对照之间的 lncRNA 处于显着差异水平(调整后的p < 0.05,|log 2 FC| ≥ 1,FC:倍数变化),而火山图则显示TSCC 之间所有 lncRNA 的FC 和p值和正常控制。在火山图中,红点代表显着上调的lncRNA,而绿点代表显着下调的lncRNA(调整后的p < 0.05,|log 2足球俱乐部| ≥1)。黑点代表 TSCC 中无显着变化的 lncRNA(图2B)。
TSCC ( n = 149) 和正常 ( n = 15) 组织样本之间差异表达 lncRNA 的热图 ( A ) 和火山图 ( B )。热图表明 TSCC 和正常对照之间的 lncRNA 处于显着差异水平(调整后的p < 0.05,|log 2 FC| ≥ 1,FC:倍数变化),而火山图则显示TSCC 之间所有 lncRNA 的FC 和p值和正常控制。在火山图中,红点代表显着上调的lncRNA,而绿点代表显着下调的lncRNA(调整后的p < 0.05,|log 2足球俱乐部| ≥1)。黑点代表 TSCC 中无显着变化的 lncRNA(图2B)。免疫相关特征lncRNA的鉴定及六种lncRNA预后模型的构建
缺少生存数据或生存时间少于 30 天的患者被排除在模型构建之外。因此,本研究纳入了 145 例 TSCC 患者进行后续分析。145 例 TSCC 患者的人口统计数据,包括年龄、性别和 TNM 分期

使用R软件,将TSCC受试者按2:1的比例随机分为训练组和测试组。在训练队列中,对 695 个差异表达的免疫相关 lncRNA 的表达谱进行单变量分析,得到 10 个具有潜在预后价值的免疫相关特征 lncRNA

对于10个免疫相关特征lncRNA,进行逐步多元Cox回归分析,进一步筛选免疫相关lncRNA,得到6个免疫相关特征lncRNA(表3)。然后使用这 6 个特征 lncRNA 构建 TSCC 的预后模型。使用第 2.4 节中描述的以下公式,计算每位患者的风险评分。然后根据中位风险评分将 TSCC 患者分为低风险组和高风险组。六个 lncRNA 基因中每个基因的p值均 < 0.05,表明这些基因具有统计显着性。

六种免疫相关 lncRNA 预后模型的验证
使用上述公式计算出训练队列患者的风险评分后,即可计算出中位风险评分并用于将患者分为高风险组和低风险组。高风险和低风险患者组之间的生存状态分布。 Kaplan-Meier 生存曲线显示高风险组和低风险组患者的生存概率存在相对明显的差异。然后,我们通过执行相同类型的分析,在测试队列中验证了模型,数据分析显示了与训练队列相似的结果
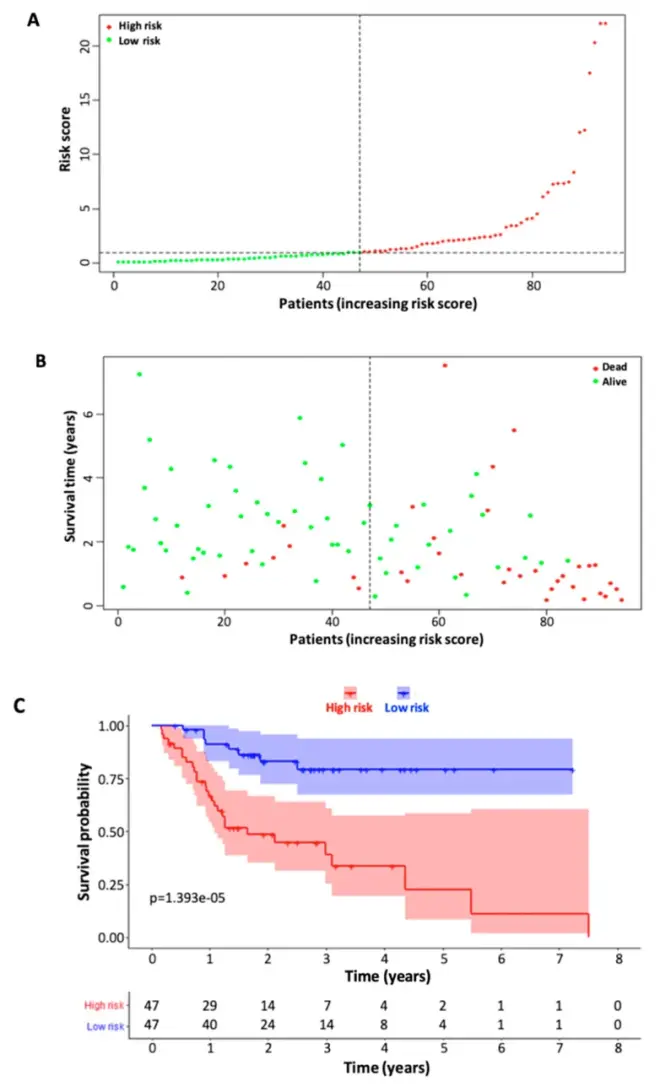 通过训练队列构建六种免疫相关lncRNA特征模型(n = 94)。( A ) 训练队列中 TSCC 患者的风险评分分布。风险评分是根据 6 个特征 lncRNA 使用以下公式计算的: 风险评分 = βgene 1 × exprgene 1 + βgene 2 × exprgene 2 + … + βgene n × exprgene n(βgene n是系数,exprgene n是表达式等级)。计算每位 TSCC 患者的风险评分,然后根据中位风险评分将患者分为高风险组和低风险组。(B)高危组和低危组(训练队列)患者的生存状态和时间。( C )高危组和低危组患者的Kaplan-Meier生存曲线。
通过训练队列构建六种免疫相关lncRNA特征模型(n = 94)。( A ) 训练队列中 TSCC 患者的风险评分分布。风险评分是根据 6 个特征 lncRNA 使用以下公式计算的: 风险评分 = βgene 1 × exprgene 1 + βgene 2 × exprgene 2 + … + βgene n × exprgene n(βgene n是系数,exprgene n是表达式等级)。计算每位 TSCC 患者的风险评分,然后根据中位风险评分将患者分为高风险组和低风险组。(B)高危组和低危组(训练队列)患者的生存状态和时间。( C )高危组和低危组患者的Kaplan-Meier生存曲线。 通过测试队列验证六种免疫相关 lncRNA 特征模型(n = 51)。( A ) 测试队列中患者的风险评分分布。( B )高危组和低危组(测试队列)患者的生存状态和时间。( C )高危和低危患者组的Kaplan-Meier生存曲线。
通过测试队列验证六种免疫相关 lncRNA 特征模型(n = 51)。( A ) 测试队列中患者的风险评分分布。( B )高危组和低危组(测试队列)患者的生存状态和时间。( C )高危和低危患者组的Kaplan-Meier生存曲线。ROC分析
基于训练队列的数据计算了六种免疫相关特征lncRNA的3年和5年ROC曲线,以评估六种lncRNA模型的性能,得出AUC值分别为0.773和0.795。对两个测试队列进行了相同的分析,其 3 年和 5 年 AUC 值分别为 0.677 和 0.754,整个队列的 AUC 值分别为 0.677 和 0.754
 六种免疫相关lncRNA特征模型的ROC分析。( A ) 训练队列 ( n = 94) 的 3 年和 5 年生存 ROC 曲线。( B ) 测试队列 ( n = 51) 的 3 年和 5 年生存 ROC 曲线。( C ) 整个队列的 3 年和 5 年生存 ROC 曲线 ( n = 125)。AUC:曲线下面积。
六种免疫相关lncRNA特征模型的ROC分析。( A ) 训练队列 ( n = 94) 的 3 年和 5 年生存 ROC 曲线。( B ) 测试队列 ( n = 51) 的 3 年和 5 年生存 ROC 曲线。( C ) 整个队列的 3 年和 5 年生存 ROC 曲线 ( n = 125)。AUC:曲线下面积。6-lncRNA模型与TSCC其他临床病理参数的比较
我们进行了单变量和多变量 Cox 回归分析,以确定我们的模型是否可以独立用于 TSCC 的预后。我们纳入了其他临床病理学特征,例如年龄、性别和分期进行比较。单变量和多变量 Cox 回归分析均表明,与临床病理因素(年龄、性别和分期)相比,我们的 6-lncRNA 模型具有较高的中位风险比和较小
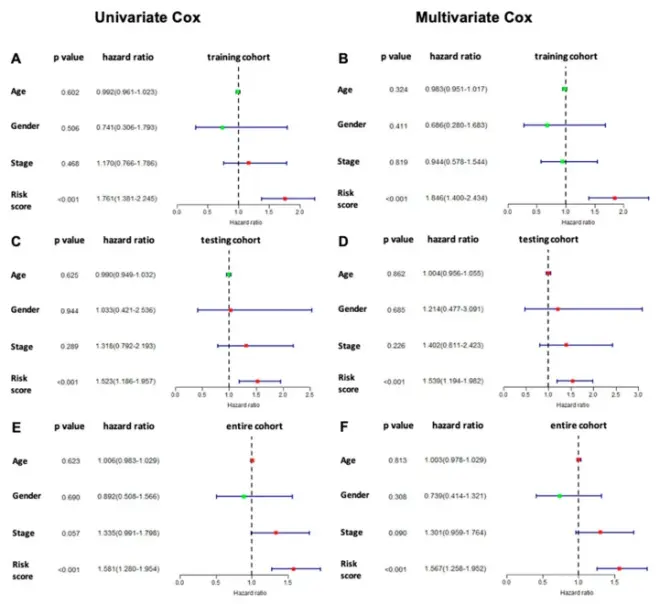 用于评估 6-lncRNA 特征模型与个体临床病理因素的单变量和多变量 Cox 回归分析。训练队列中 6-lncRNA 模型的( A ) 单变量和 ( B ) 多变量 Cox 回归分析。测试队列中 6-lncRNA 模型的( C ) 单变量和 ( D ) 多变量 Cox 回归分析。( E ) 单变量和 ( F)对整个队列中的 6-lncRNA 模型进行多变量 Cox 回归分析。单变量 cox 回归独立于所有其他变量计算变量与 TSCC 患者生存之间的风险比,而多变量 cox 回归在计算风险比时考虑存在的其他变量。与临床病理因素、年龄、性别和癌症分期相比,我们的 6-lncRNA 模型具有较高的中位风险比和较小的变异。
用于评估 6-lncRNA 特征模型与个体临床病理因素的单变量和多变量 Cox 回归分析。训练队列中 6-lncRNA 模型的( A ) 单变量和 ( B ) 多变量 Cox 回归分析。测试队列中 6-lncRNA 模型的( C ) 单变量和 ( D ) 多变量 Cox 回归分析。( E ) 单变量和 ( F)对整个队列中的 6-lncRNA 模型进行多变量 Cox 回归分析。单变量 cox 回归独立于所有其他变量计算变量与 TSCC 患者生存之间的风险比,而多变量 cox 回归在计算风险比时考虑存在的其他变量。与临床病理因素、年龄、性别和癌症分期相比,我们的 6-lncRNA 模型具有较高的中位风险比和较小的变异。 六种免疫相关lncRNA模型的预后价值
进行 ROC 分析以确定 6 个 lncRNA 特征与其他临床病理因素(如年龄、性别、分期、T 和 N)相比的临床意义。ROC 曲线表明,与其他因素相比,我们的 6-lncRNA 模型在 3 年和 5 年总生存率
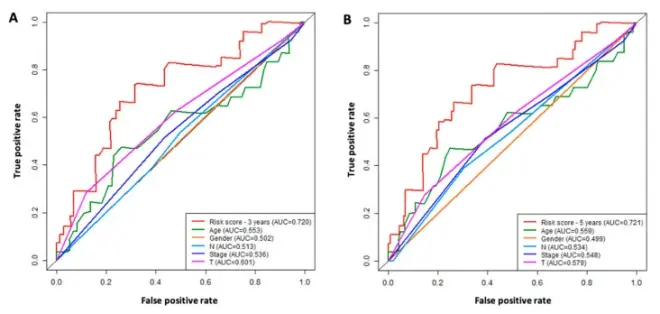 ROC 分析用于比较 6-lncRNA 特征模型与临床病理因素。( A ) 风险评分(基于六-lncRNA模型)和临床病理因素(年龄、性别、分期、淋巴结N、肿瘤大小T)的3年生存ROC曲线。( B ) 风险评分(基于六-lncRNA模型)和临床病理因素(年龄、性别、分期、淋巴结N、肿瘤大小T)的5年生存ROC曲线。六-lncRNA 特征模型显示了最高的 AUC 值。
ROC 分析用于比较 6-lncRNA 特征模型与临床病理因素。( A ) 风险评分(基于六-lncRNA模型)和临床病理因素(年龄、性别、分期、淋巴结N、肿瘤大小T)的3年生存ROC曲线。( B ) 风险评分(基于六-lncRNA模型)和临床病理因素(年龄、性别、分期、淋巴结N、肿瘤大小T)的5年生存ROC曲线。六-lncRNA 特征模型显示了最高的 AUC 值。与六种特征 lncRNA 相关的免疫状态
基于六种免疫相关特征lncRNA或所有免疫相关lncRNA的表达谱进行PCA分析以显示高风险组和低风险组之间的差异。根据六种免疫相关特征 lncRNA 的表达谱,两个患者组之间存在明显的区别。两个患者组倾向于不同方向分布,表明高风险和低风险患者组之间的免疫状态存在显着差异。
 主成分分析(PCA)。( A ) 基于所有免疫相关 lncRNA 的表达谱对高风险和低风险患者组进行 PCA。( B ) 基于六种免疫相关特征 lncRNA 表达谱的高风险和低风险患者组之间的 PCA。根据六种免疫相关特征 lncRNA 的表达谱,高风险和低风险患者组被很好地分开。
主成分分析(PCA)。( A ) 基于所有免疫相关 lncRNA 的表达谱对高风险和低风险患者组进行 PCA。( B ) 基于六种免疫相关特征 lncRNA 表达谱的高风险和低风险患者组之间的 PCA。根据六种免疫相关特征 lncRNA 的表达谱,高风险和低风险患者组被很好地分开。
标签:













